La diarrea es una de las principales causas de mortalidad neonatal, disminución de ganancia de peso y aumento del uso de medicamentos en los cerdos. Los principales patógenos son bacterias como Escherichia coli (E. coli) y Clostridium perfringens (C. perfringens), así como Rotavirus y Coronavirus.
Un estudio realizado a partir de muestras de un programa de control en diferentes países europeos evaluó la presencia de estos patógenos en las heces de lechones con diarrea neonatal en el año 2020.

Material y métodos
Todas las muestras utilizadas en este estudio procedían de lechones lactantes. Se analizó un total de 324 muestras, la mayoría fecales, procedentes de 116 granjas para detectar E. coli, C. perfringens y Clostridioides difficile (C. difficile). Las muestras de 79 de estas granjas se examinaron además mediante PCR para detectar Rotavirus del grupo A y C, así como para detectar el virus de la Diarrea Epidémica Porcina (PEDV) y el virus de la Gastroenteritis Transmisible (TGEV). Por lo tanto, el estudio consistió en dos análisis, basados en esta diferencia en el espectro de patógenos examinados en las granjas. En el primer análisis (1), se evaluó la frecuencia de las bacterias patógenas en todas las muestras de las 116 granjas. En el segundo análisis (2), se examinaron los datos combinados de bacterias y virus en 79 de las granjas para detectar simultáneamente más de un patógeno causante de diarrea neonatal.
La mayoría de las 116 granjas (figura 1) estaban situadas en Alemania (DE; n=59), seguidas de los Países Bajos (NL; n=19) y Polonia (PL; n=19), Dinamarca (DK; n=9), Reino Unido (UK; n=8), Austria (AU, n=1) e Irlanda (IR; n=1).

Figura 1. Número de granjas participantes por país de origen.
Los cultivos aislados de E. coli y C. perfringens en ambos análisis se tipificaron a nivel molecular para determinar la presencia de genes asociados a virulencia y así evaluar su potencial virulento (Strutzberg-Minder and Dohmann 2015). Además, en el análisis 1 se analizó el contenido bacteriano semicuantitativo de E. coli, C. perfringens y C. difficile en función de su crecimiento en cultivo (tabla. 1).
Resultados
Análisis 1:
En el examen bacteriológico de las muestras de las 116 granjas, se encontraron un total de 710 aislados (figura 2). Las bacterias más frecuentemente aisladas fueron E. coli (48,6%), seguida de C. perfringens (33,9%) y C. difficile (15,9%). El análisis semicuantitativo mostró un crecimiento moderado o alto para el 99,4% de los aislados de E. coli y el 96,7% de los aislados de C. perfringens. Sin embargo, para C. difficile se encontró un crecimiento moderado (46,9%) o bajo (53,1%). Sólo el 16% de los aislados de E. coli presentaban potencial hemolítico (tabla 1) e incluso entre los patotipos de EC definidos y los EC potencialmente virulentos (figura 3), sólo el 16% de los aislados eran hemolizantes, lo que confirma que la hemólisis no es una característica única de la virulencia.

Figura 2. Número de aislados bacterianos detectados (n total: 710) en 116 granjas por país de origen. Número de granjas por país entre paréntesis.
Tabla 1: Número (n) de aislados bacterianos detectados en cultivo en el análisis 1 y proporciones de niveles semicuantitativos dentro de cada especie bacteriana.
| n | % | |||
|---|---|---|---|---|
| contenido alto | contenido moderado | contenido menor | ||
| Escherichia coli | 345 | 77,1 | 22,3 | 0,6 |
| de estas, con potencial de hemólisis | 57 (16,5%) | |||
| Clostridium perfringens | 241 | 46,9 | 49,8 | 3,3 |
| Clostridioides difficile | 113 | 0,0 | 46,9 | 53,1 |
Tras tipificar los aislados de E. coli y C. perfringens y relacionarlos con cada granja, quedaron un total de 276 aislados de E. coli y 117 de C. perfringens. Se diferenciaban entre sí por sus factores de virulencia y sus patrones de genes de toxinas dentro de una misma granja. Basándose en su patrón de genes de virulencia, el 19,9% (n=55) de estos aislados de E. coli pudieron asignarse a uno de los patotipos conocidos relacionados con la diarrea neonatal (EDEC, EPEC, ETEC, NTEC). Otros 104 aislados (37,7%) eran portadores de varios genes de fimbrias y adhesinas, así como de toxinas, por lo que pueden clasificarse como potencialmente virulentos. Por lo tanto, un total del 57,6% de los diferentes aislados de E. coli (n=159) fueron clasificados como virulentos o potencialmente virulentos para la diarrea neonatal. La figura 3 muestra la distribución de los resultados de tipificación de E. coli por país de origen.

Figura 3. Número y distribución de los tipos de E. coli de un total de 276 aislados por país de origen. Número de aislados por país entre paréntesis.
La tipificación de los cultivos aislados de C. perfringens reveló que todos ellos pertenecían a C. perfringens tipo A (CPA). El 90,6% de ellos (n=106) portaban los genes codificantes tanto de la toxina α como de la ß2, por lo que mostraban un alto potencial de virulencia. El resto de los cultivos aislados (9,4%, n=11) sólo mostraban el gen de la α-toxina (figura 4).
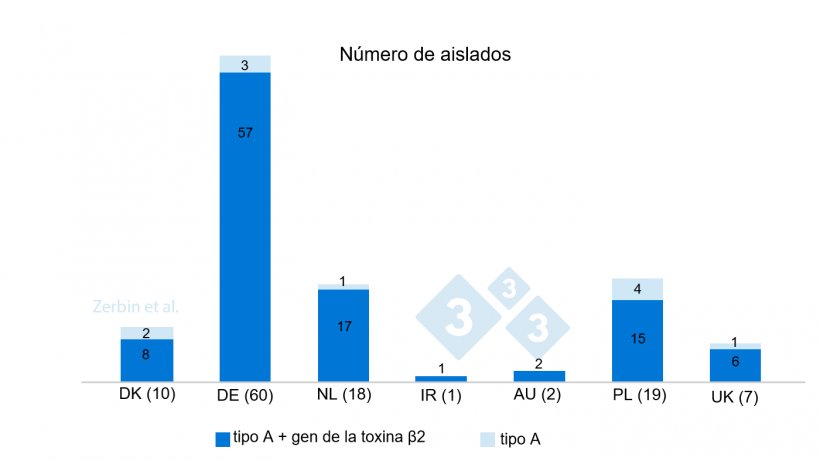
Figura 4. Número y porcentaje de aislados de C. perfringens con y sin el gen de la toxina ß2 de un total de 117 aislados por país de origen. Número de aislados por país entre paréntesis.
Análisis 2:
En cuanto al subgrupo de las 79 granjas, los patógenos encontrados con mayor frecuencia en estas fueron E. coli (96,2%), C. perfringens (87,3%), Rotavirus (74,7%) y C. difficile (46,8%). El PEDV sólo se detectó en dos granjas y el TGEV en ninguna. Por lo tanto, el grupo de Coronavirus no se consideró en el análisis posterior.
La tipificación posterior reveló aislados de E. coli virulentos o potencialmente virulentos en el 78,9% (n=60) de las granjas, y CPA con el gen de la toxina ß2 en el 81,0% (n=64).
En el 25,3% (n=20) de las granjas se encontraron simultáneamente los cuatro patógenos diferentes, tres patógenos diferentes en el 39,2% (n=31), dos patógenos en el 31,6% (n=25) y en el 3,8% (n=3) de las granjas sólo se detectó un patógeno. Sólo en una de las granjas no se encontró ninguno de los patógenos analizados.
Entre ellos, la combinación de E. coli con Rotavirus y CPA con toxina β2 se dio en la mayoría de estas granjas (n=19/ 24,1%). La combinación con E. coli, CPA y C. difficile se produjo con una frecuencia significativamente menor (n=8).

E. coli fue el patógeno encontrado más frecuentemente, se presentó principalmente en combinación con CPA con el gen de la toxina β2 (64,6%) o con rotavirus (64,6%).
Discusión
El presente estudio analizó la aparición de patógenos relevantes causantes de diarrea neonatal en granjas europeas que participaron voluntariamente en un programa de seguimiento de diarrea en lechones. La interpretación de los presentes resultados debe hacerse con cuidado, ya que existe un cierto sesgo de selección en el diseño de la muestra. Las granjas enviaron las muestras voluntariamente, ya que presentaban problemas de diarrea. No obstante, los datos muestran tendencias realistas y claras en relación con los patógenos causantes de la diarrea neonatal que ya se han descrito en la literatura.
Los dos análisis de este estudio confirman que E. coli y C. perfringens son los principales patógenos bacterianos que causan diarrea a los lechones lactantes. Los datos actuales mostraron que casi el 58% de los aislados de E. coli y casi el 91% de los de C. perfringens pueden considerarse virulentos o potencialmente virulentos. En el segundo análisis, los aislados de E. coli y C. perfringens pudieron encontrarse en aproximadamente el 80% de las granjas.
Un resultado interesante es la muy baja evidencia de Coronavirus (PEDV y TGEV) en las muestras del estudio. Aunque se mencionan como posibles agentes de la diarrea neonatal, se describen poco en la literatura y la diarrea causada por el TGEV se ha vuelto poco común en Europa. El análisis combinado de los diferentes patógenos bacterianos y víricos en las 79 granjas reveló hallazgos simultáneos de dos o más patógenos en casi el 95% de las granjas. Esto demuestra que la diarrea neonatal en los lechones rara vez tiene una etiología monocausal. Esto debe ser considerado con en el momento de tomar decisiones respecto a medidas profilácticas y terapéuticas.



