Los virus de la influenza porcina (SIV; siglas en inglés) pertenecen al género Influenzavirus tipo A de la familia Orthomyxoviridae. Estos virus se subclasifican en base a las glicoproteínas de superficie: la hemaglutinina (HA) y la neuraminidasa (NA), de las cuales se describen 17 y 10 tipos, respectivamente. La combinación de un tipo de HA con un tipo de NA es lo que define a lo que conocemos como subtipo. Así, los subtipos mayoritarios en el cerdo son el H1N1, el H1N2 y el H3N2.
En base a los distintos estudios realizados en Europa, podemos afirmar que la seroprevalencia de los tres subtipos (H1N1, H1N2 y H3N2) es muy alta en los países con una producción porcina importante. Por ejemplo, en Alemania, Bélgica, Dinamarca o España, la seroprevalencia de los tres subtipos es superior al 30%. Concretamente en España, el 94% de las explotaciones son positivas al menos a un subtipo. En Francia o Reino Unido se detectan elevadas prevalencias de H1N1 o H1N2; sin embargo, parece que el subtipo H3N2 no estaría presente al menos durante los últimos años. En países con una producción menor, como por ejemplo la República Checa, Irlanda o Polonia, la prevalencia de los SIV parece ser inferior.

En una misma explotación, es posible detectar de forma directa (mediante PCR) o de forma indirecta (mediante inhibición de la hemaglutinación) más de un subtipo de SIV circulante. De hecho, esta situación, ha sido demostrada en explotaciones de Holanda, Francia o España, entre otros. Individualmente, cabe añadir que la proporción de animales que presentan anticuerpos frente a más de un subtipo puede llegar a ser elevada tanto en cerdas reproductoras como en cerdos de engorde. Este hecho sugiere que la infección por SIV es activa y frecuente. A pesar de ello, la presencia del virus no siempre está asociada a brotes de patología respiratoria; de hecho, existe un elevado número de explotaciones positivas al virus sin clínica aparente. En definitiva, estos hallazgos sugieren que SIV se presenta de manera importante como una infección enzoótica.
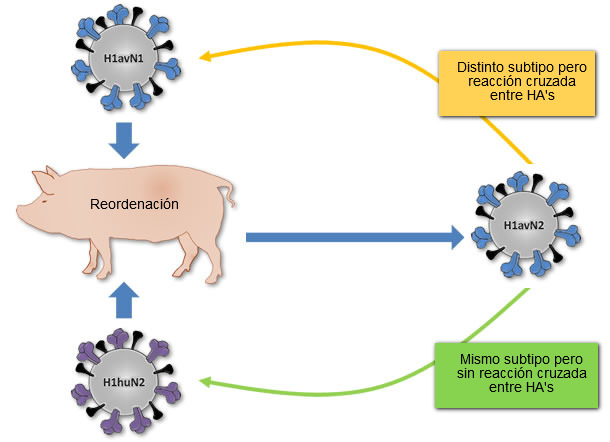
Figura 1. Origen del virus H1avN2 descrito en Dinamarca y Francia. Este virus se genera a partir de la reordenación de los virus H1avN1 (que aporta la HA) y el virus H1huN2 (que aporta la NA). Al igual que el virus H1huN2, el H1avN2 virus pertenece al subtipo H1N2, pero su HA no reaccionará de forma cruzada con este virus, mientras que si lo hará con la HA del virus H1avN1 aunque este pertenezca a un subtipo distinto (H1N1).
La clasificación por subtipos es útil para poder simplificar y entender la epidemiologia y diversidad de los virus de la influenza; sin embargo, lo cierto es que la diversidad de los SIV es más compleja. Cómo muy bien resume la Dra. Simon en otro artículo publicado en esta misma página web, en Europa se pueden distinguir como mínimo 4 linajes principales en función del tipo de HA que contengan. Así, distinguiríamos los relacionados con los H1N1 tipo aviar (H1avN1), los relacionados con los H1N2 (H1huN2), los relacionados con los H1N1 pandémico (H1N1pdm) y finalmente los relacionados con los H3N2.
Es necesario recordar que los SIV, al igual que cualquier otro virus perteneciente a los influenzavirus A, están compuestos por un genoma segmentado en 8 fragmentos de RNA lo cual permite que evolucionen mediante dos mecanismos: 1) la deriva antigénica; mutaciones que cuando se producen en lugares específicos de la HA (principalmente) o la NA pueden generar variantes víricas que escapen de la inmunidad generada previamente en la población por virus ancestros (causa principal de la gripe estacional en el ser humano), y 2) la reordenación genética, la cual se produce cuando dos o más virus intercambian fragmentos de RNA (por ejemplo, la generación en el año 1994 de la cepa H1huN2, resultante de la reordenación entre un H1N1 humano y el H3N2 porcino).
A pesar de que los 3 H1 distintos (H1avN1, H1huN2, y H1N1pdm) han tenido un ancestro común en algún momento, no cruzan a nivel antigénico o lo hacen de forma limitada. Probablemente, esto se deba a que las tres H1 han evolucionado por deriva antigénica de forma aislada en tres especies distintas antes de introducirse en el cerdo en Europa; en aves el H1avN1, en humanos el H1huN2, y en cerdos en Norteamérica el H1N1pdm. Por otro lado, la reordenación genética puede generar nuevas cepas de virus de la influenza que, aun perteneciendo a un subtipo, pueden cruzar antigénicamente con virus pertenecientes a otros subtipos. Un claro ejemplo de ello es la cepa H1avN2 descrita en Dinamarca y en Francia. Dicha cepa contiene una HA relacionada con la presente en las cepas H1avN1 y en consecuencia no cruza antigénicamente con la cepa H1huN2, pero sí lo hace con las cepas H1avN1.
La diversidad genética y antigénica de los virus de la influenza debe entenderse como algo dinámico y en evolución constante. Los trabajos realizados en Alemania, Dinamarca, España o Italia, recientemente publicados, así lo demuestran. En dichos trabajos se han aislado nuevas cepas producto de la reordenación entre las cepas de SIV porcinas y cepas de la gripe estacional humana. Por todas estas razones, es de vital importancia mantener e incluso fomentar una vigilancia epidemiológica activa de SIV con el objeto de conocer las cepas presentes en la cabaña porcina así como su prevalencia e impacto.



