La diversidad de los virus de la influenza se explica principalmente por dos aspectos muy relacionados entre sí. Por un lado, los mecanismos evolutivos principales a los que este virus está sometido —la deriva y la reordenación o intercambio genético (discutidos en otro artículo de esta misma sección)—; por otro, el comportamiento epidemiológico del virus en cada especie, el cual, como discutiremos en el presente artículo, tiene un importante efecto sobre los mecanismos evolutivos.
Si comparamos en base a la deriva genética los virus presentes en el cerdo con los presentes en el ser humano, caben destacar principalmente dos fenómenos; 1) dada una cepa concreta, el virus en el ser humano varía antigénicamente (cambios en amino ácidos) más deprisa que en el cerdo, aunque la tasa de mutación genética del virus es igual para ambas especies, y 2) a nivel global, la diversidad del virus de la influenza, tanto a nivel genético como antigénico, es mucho mayor en cerdos que en el ser humano. A pesar de que estos dos hechos pueden parecer a priori contradictorios, son, como veremos a continuación, perfectamente compatibles.

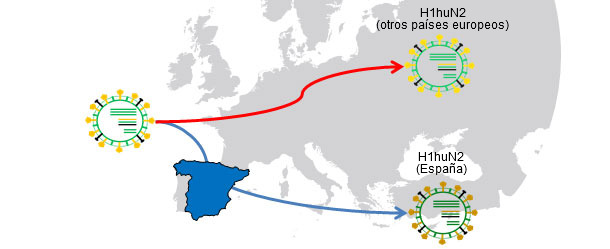
Figura 1. Ancestro común H1huN2 (1994); Aislamiento geográfico → la deriva produce variantes relacionadas con cada país.
En otros artículos de esta misma sección hemos visto que la influenza en el cerdo se puede comportar de forma endémica o enzoótica, dicho de otro modo, que los virus de la influenza pueden "residir" en las explotaciones. Esta situación es muy frecuente y supone la aparición de circulaciones recurrentes. En un trabajo del año 2001 (de Jong y colaboradores) se compararon genética y antigénicamente más de 20 H1N1 porcinos de "tipo aviar" (H1avN1). Estos virus habían sido aislados en 6 explotaciones de Holanda que estaban endémicamente infectadas. En base al gen de la hemaglutinina (H), se comprobó que los aislados formaban cinco grupos que se diferenciaban tanto a nivel genético cómo antigénico. Cabe destacar que cada grupo correspondía a una granja (salvo uno de los grupos que incluía aislados de dos explotaciones). Este trabajo demuestra que todos los aislados tienen un H1avN1 ancestro común pero que, gracias a la circulación endémica, los virus evolucionan y se diferencian de forma aislada. En otro estudio (Martín-Valls et al., 2013), realizado en el CReSA entre los años 2009 y 2012, se aislaron 4 virus H1N2. Al analizar la H1 desde un punto de vista genético, se observó que todos ellos pertenecían al tipo H1N2 "tipo humano" (H1huN2), pero que formaban un grupo claramente diferenciado dentro de los H1huN2; podía concluirse que había existido una evolución aislada en España de este tipo. Posteriormente, hemos podido comprobar que estos virus reaccionan de forma limitada con sueros que si reaccionaban con otro H1huN2 comercial, indicando cierta deriva antigénica (datos no publicados). En definitiva, tanto los factores epidemiológicos como la evolución por aislamiento en explotaciones en situaciones endémicas, o el aislamiento geográfico, juegan un papel importante en la aparición de nuevas variantes antigénicas. En el ser humano, sin embargo, la situación es completamente distinta. En este caso podríamos decir que, debido a la presión inmunitaria global, los virus están forzados a cambiar antigénicamente para poder sobrevivir. Actualmente, las múltiples y variadas conexiones que existen entre países de todo el mundo eliminan las situaciones de aislamiento. Por tanto, en el ser humano existe una selección positiva global de aquellas variantes de virus de la influenza capaces de escapar de la respuesta inmune; este fenómeno se traduce en la necesidad constante de una actualización de las vacunas en el ser humano.
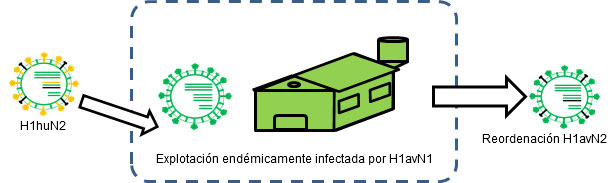
Figura 2. H1huN2 se transmite a una granja endémicamente infectada por H1avN1 → Se produce reordenación.
La reordenación o intercambio genético también puede ser origen de cambios a nivel antigénico, incluso de una forma más súbita. En el ser humano, por ejemplo, ésta ha sido la causa principal de las últimas 3 pandemias ya que ha supuesto la introducción de, al menos, una nueva hemaglutinina distinta antigénicamente a la pre-existente en la población y frente a la cual no había inmunidad previa. En otro artículo de esta sección ya vimos como en el cerdo los virus de la influenza porcina sufren reordenaciones de forma frecuente, generándose en consecuencia nuevas cepas. Ejemplos de ello son el H1avN2, un H1N2 que cruza antigénicamente con los virus H1avN1 (Rose et al., 2013), o los H1huN1, que cruzan antigénicamente con el H1huN2 (Martín-Valls et al., 2013; Rose et al., 2013). Para que estas reordenaciones ocurran es necesario que dos cepas distintas infecten un mismo animal al mismo tiempo. Obviamente, las situaciones endémicas son escenarios ideales para que esto ocurra. De hecho, cuando se han evaluado situaciones endémicas, se han observado evidencias de reordenación genética entre distintas cepas e incluso subtipos (Martín-Valls et al., 2013; Rose et al., 2013).
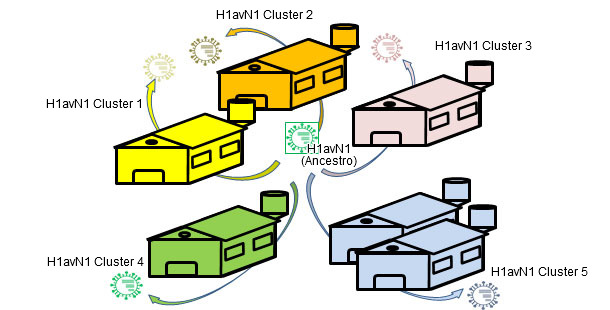
Figura 3. Las infecciones endémicas facilitan la evolución aislada por deriva en cada granja → heterogenicidad genética y antigénica relacionada con la granja.
En conclusión, los virus de la influenza porcina presentan una alta diversidad genética y antigénica generada por los mecanismos evolutivos intrínsecos de los virus. Dicha diversidad está potenciada por factores epidemiológicos como las situaciones de circulación endémica y/o el aislamiento geográfico, viéndose acentuada a lo largo del tiempo. La interacción de todos estos factores puede tener implicaciones en la falta de reconocimiento inmunológico; es decir, en un déficit en la protección cruzada entre cepas pertenecientes a un mismo subtipo.



